PI025/CE v009.00/2019-04-30 (H001 v3 / H002 v2 / H023 v3 / H024 v3 / H026 v2 / H027 v3 / H037 v2 / H039 v6 / H072 v3)
Page 6 of 12
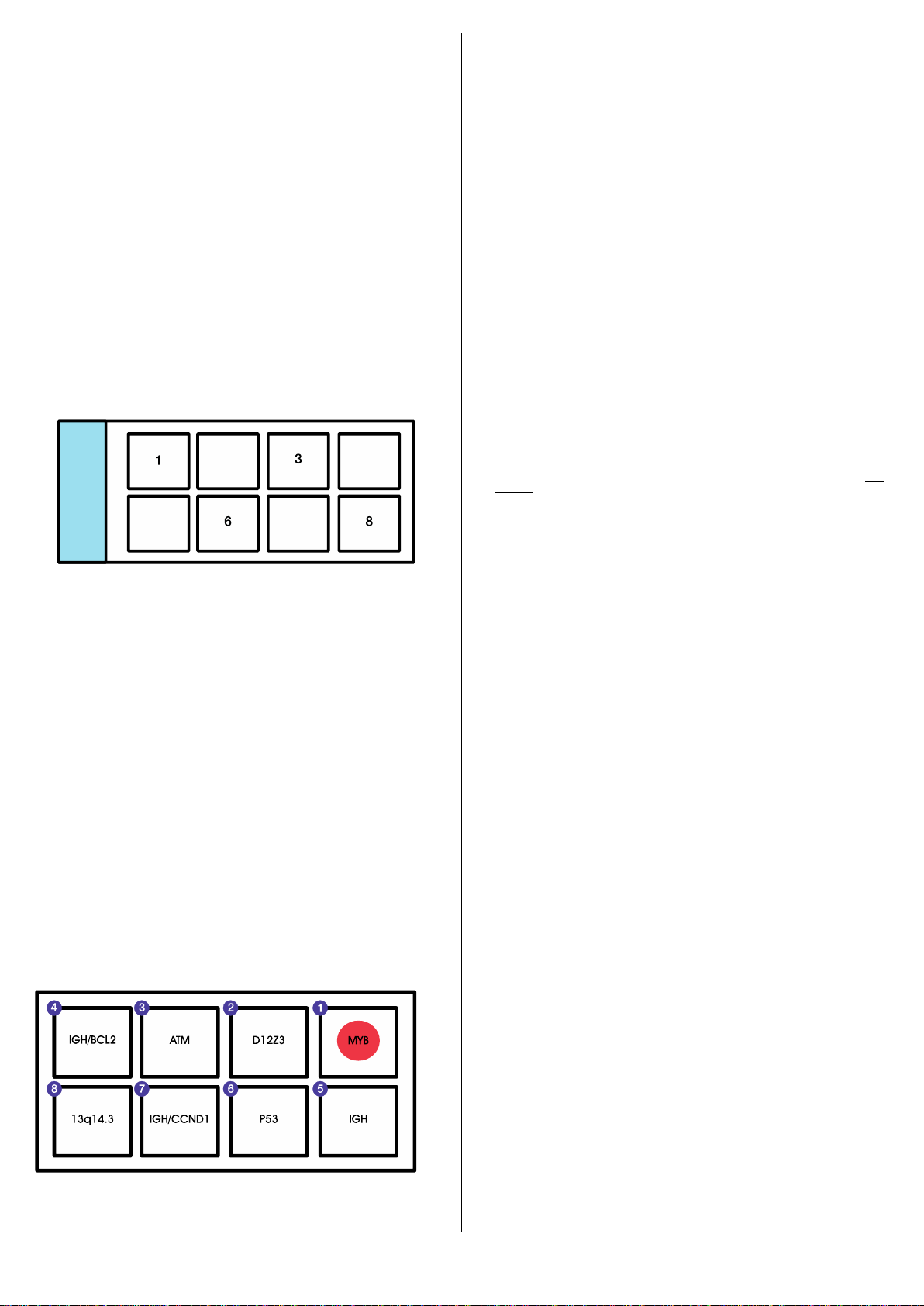
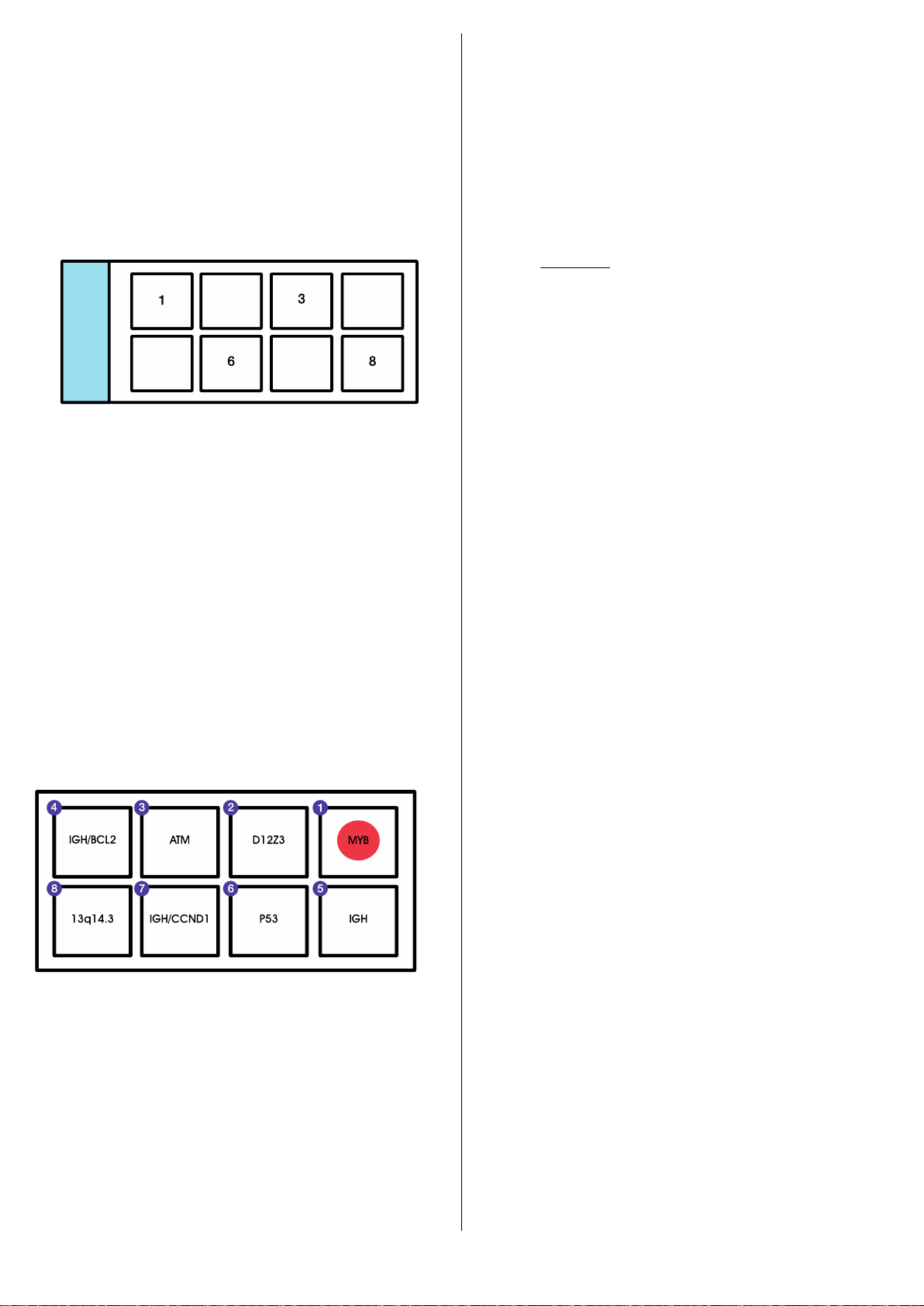
IGH/BCL2 Translocation, Dual Fusion
Dans une cellule normale, ces sondes apparaissent comme des points rouges et verts discrets,
un pour chaque chromosome homologue (entraînant une conformation 2R, 2V). Chez un patient
t(14;18)(q32.3;q21), deux signaux de fusion jaunes devraient être observés en plus des signaux
rouges et verts des chromosomes normaux 18 et 14 respectivement (1R, 1V, 2J).
IGH Breakapart
Dans une cellule normale, deux signaux rouges/verts (ou de fusion jaunes) sont attendus (2J).
Dans une cellule comportant une translocation IGH monoallélique, un signal rouge distinct et un
signal vert doivent être observés en plus d'un signal rouge/vert (ou de fusion jaune) du
chromosome 14 normal (1R, 1V, 1J). Dans le cas d'une translocation biallélique, aucun signal
de fusion n'est présent, deux signaux rouges et deux signaux verts distincts doivent être
observés (2R, 2V).
P53 Deletion
Dans une cellule normale, deux signaux rouges et verts (2R, 2V) doivent être observés alors
qu'une cellule comportant une délétion P53 doit présenter un signal rouge et deux signaux verts
(1R, 2V).
IGH/CCND1 Translocation, Dual Fusion
Dans une cellule normale, ces sondes apparaissent comme des points rouges et verts discrets,
un pour chaque chromosome homologue (entraînant une conformation 2R, 2V). Chez un patient
t(11;14)(q13;q32.3), deux signaux de fusion jaunes devraient être observés en plus des signaux
rouges et verts des chromosomes normaux 11 et 14 respectivement (1R, 1V, 2J).
13q14.3 Deletion
Une cellule normale doit présenter deux signaux rouges et deux signaux verts (2R, 2V). Une
cellule comportant une délétion hémizygote de 13q14.3 doit présenter un signal rouge et deux
signaux verts (1R, 2V) alors que deux signaux verts, sans aucun signal rouge, (0R, 2V) doivent
être observés pour une cellule comportant une délétion homozygote.
Limitations
Le signalement et l'interprétation des résultats FISH doivent être conformes aux normes de
pratique professionnelle et doivent tenir compte des autres informations cliniques et
diagnostiques. Ce kit tient lieu de succédané aux autres tests diagnostics de laboratoire et
aucune action thérapeutique ne doit être initiée sur la base des résultats FISH uniquement.
Si le protocole n’est pas respecté, la performance peut être affectée et des résultats
erronés/positifs/négatifs peuvent être obtenus.
Ce kit n'a pas été validé pour une utilisation en dehors du cadre prévu et indiqué.
Informations supplémentaires
Pour plus d’informations sur le produit, veuillez contacter l’Assistance technique Cytocell.
T: +44 (0)1223 294048
W: www.cytocell.com
ITALIANO
Introduzione
L'ibridazione in situ in fluorescenza (Fluorescence In Situ Hybridisation - FISH) è una tecnica che
permette di rilevare sequenze di DNA su cromosomi in metafase o in nuclei in interfase di
campioni citogenetici fissati, o in coltura dopo prelievo. La tecnica prevede l'utilizzo di sonde di
DNA in grado di ibridare con l'intero cromosoma o con singole sequenze. La FISH costituisce
quindi un potente strumento in aggiunta alle tecniche citogenetiche classiche. Recenti sviluppi
hanno reso possibile che questa preziosa tecnica può ora essere applicata come strumento
diagnostico essenziale nell’analisi cromosomica prenatale, ematologia e patologica. Il DNA
bersaglio, dopo la fissazione, è sottoposto a denaturazione al calore in presenza di formamide.
Il DNA bersaglio è così disponibile per l’annealing con una sonda di DNA a singola elica a
sequenza complemetare, marcata con una sostanza fluorescente. Terminata l’ibridazione, la
sonda di DNA non legata o legata in modo non specifico, è rimossa per mezzo di lavaggi
stringenti ed il DNA è in seguito colorato con un colorante di contrasto. L’ibridazione della sonda
viene infine analizzata con un microscopio a fluorescenza.
Informazioni sulle sonde
Chromoprobe Multiprobe ®CLL Cytocell è stata sviluppata per consentire alle sonde di rilevare
del (6q), trisomia 12, del (13q), del (17p), del (11q) e riarrangiamenti di IGH, il che porta a
marcatori 14+, da applicare contemporaneamente a un campione di paziente in una singola
reazione FISH. Le sonde sono state sviluppate per dare risultati chiari in cellule che non si
dividono e portare così al massimo le informazioni ricevute dalla tecnica. Il dispositivo può essere
utilizzato per diagnosi iniziale, per la conferma di risultati di citogenetica di routine e anche per
monitorare il paziente nel tempo. Il dispositivo ha anche un set di sonde per t(11;14) per
consentire ai pazienti con anomalia CLL di essere distinti da pazienti con linfoma mantellare.
Specifiche della sonda
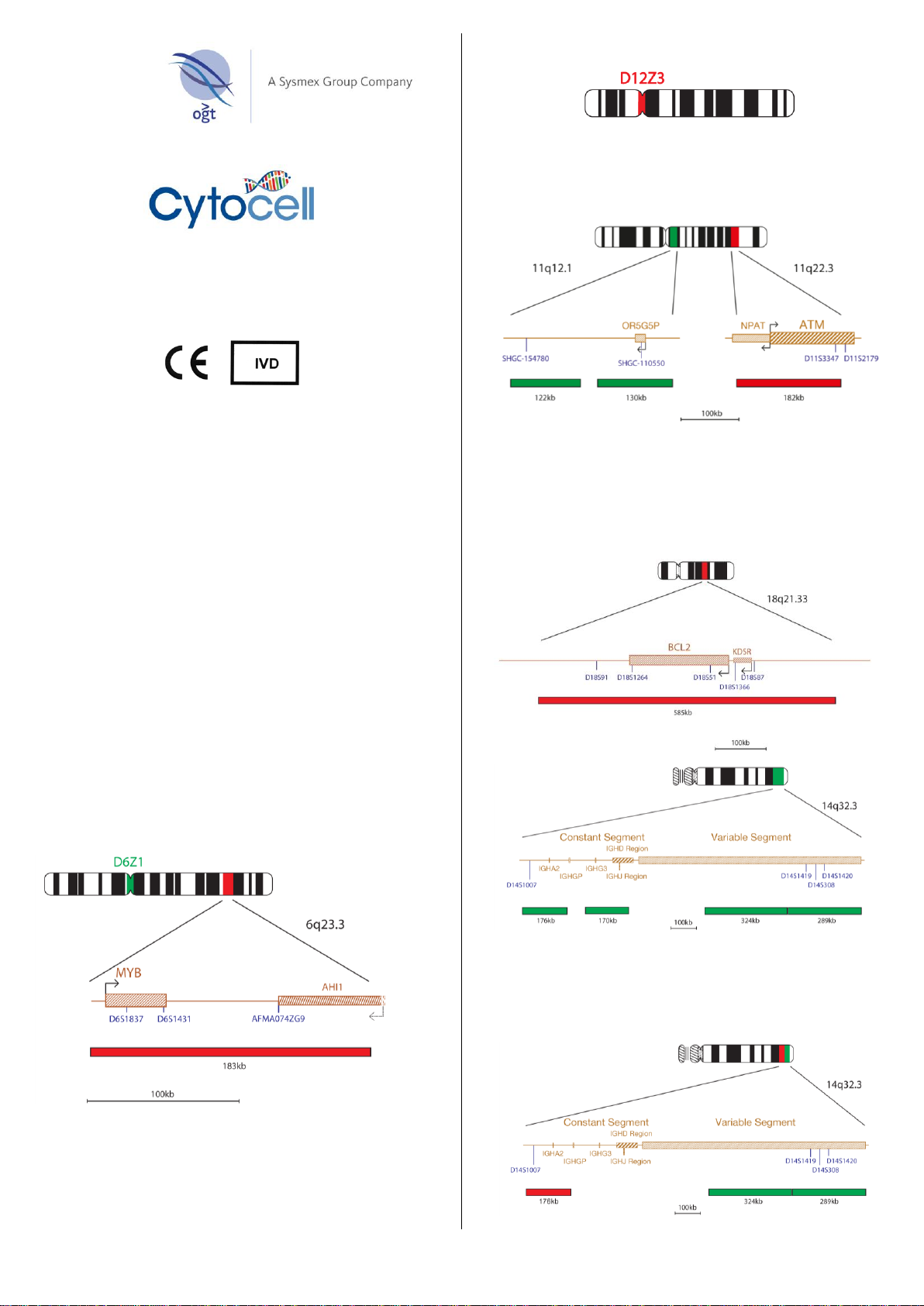
Regione MYB, 6q23.3 rosso (5.25-8.85ng/test)
Regione D6Z1, 6p11.1-q11.1 verde (6.85-10.3ng/test)
La sonda MYB è di 183kb, marcata in rosso, copre tutto il gene MYB e una regione telomerica
rispetto al gene, 137kb oltre il marcatore AFMA074ZG9. Il mix della sonda contiene anche una
sonda di controllo per il centromero 6 (D6Z1), marcata in verde.
Chromosome 12 Enumeration
Regione D12Z3, 12p11.1-q11.1 rosso (0.28-0.47ng/test)
La sonda alfa satellite per il cromosoma 12 è una sonda a sequenza ripetuta, marcata in rosso,
che riconosce la sequenza centromerica ripetuta D12Z3.
ATM Deletion
Regione ATM, 11q22.3 rosso (5.25-8.85ng/test)
Regione 11q12.1 verde (20.6-30.8ng/test)
La sonda ATM è lunga 182kb, marcata in rosso, e copre l'estremità telomerica del gene NPAT e
l'estremità centromerica del gene ATM subito oltre il marcatore D11S3347. Il mix della sonda
contiene 11q12.1 sonda di controllo marcata in verde, che copre una zona di 122kb compreso il
marcatore SHGC-154780 e una Regione 130kb compreso il marcatore SHGC-110550.
IGH/BCL2 Translocation, Dual Fusion
Regione BCL2, 18q21.33 rosso (5.60-9.44ng/test)
Regione IGH, 14q32.33 verde (13.7-20.5ng/test)
Il prodotto IGH/BCL2 è costituito da sonde, marcate in verde, che coprono i segmenti costante,
J, D e variabile del gene IGH e da una sonda di 585kb, marcata in rosso, che copre i geni BCL2
e KDSR.
IGH Breakapart
Regione IGHC, 14q32.33 rosso (5.25-8.85ng/test)
Regione IGHV, 14q32.33 verde (13.7-20.5ng/test)
Il prodotto IGH è costituito da una sonda di 176kb, marcata in rosso, che copre la regione
costante del gene, e da una sonda verde, che copre una regione di 617kb all'interno del
segmento variabile del gene.
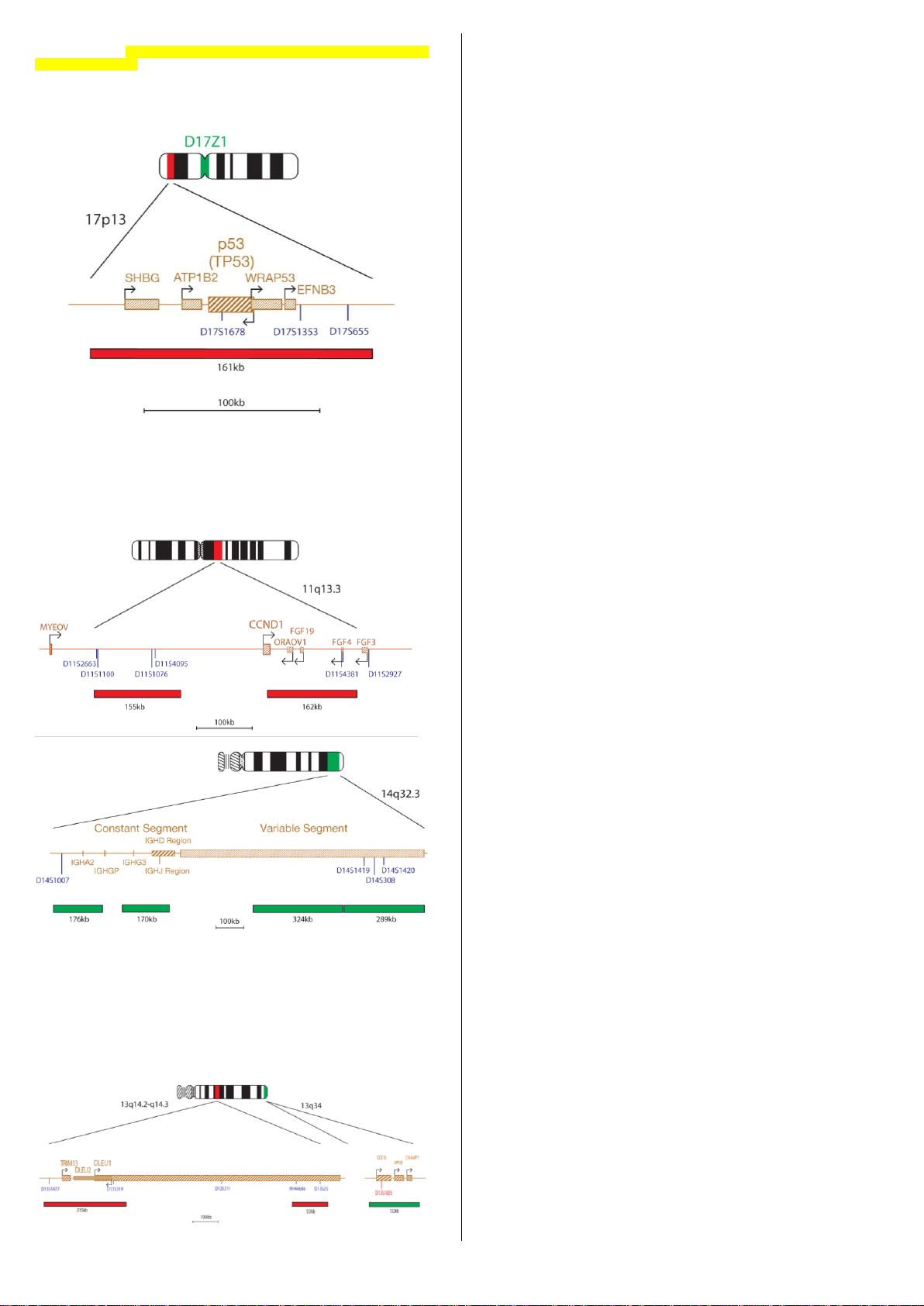
P53 Deletion
Regione P53, 17p13.1 rosso (4.20-7.08ng/test)
Regione D17Z1, 17p11.1-q11.1 verde (13.7-20.5ng/test)
La sonda P53 è di 161kb, marcata in rosso, copre tutto il gene P53 (TP53), con una lunghezza
di 66kb telomerica rispetto al gene e copre una regione centromerica rispetto al gene, fino a
subito oltre il marcatore D17S655. Il mix della sonda contiene anche una sonda di controllo per
il centromero 17 (D17Z1), marcata in verde.
IGH/CCND1 Translocation, Dual Fusion
Regione CCND1 (BCL1), 11q13.3 rosso (5.60-9.44ng/test)
Regione IGH, 14q32.33 verde (13.7-20.5ng/test)
Il prodotto IGH/CCND1 è costituito da sonde, marcate in verde, che copronoi segmenti costante,
J, D e variabile del gene IGH e da sonde CCND1, marcate in rosso. Il mix della sonda CCND1
contiene una sonda di 155kb centromerica rispetto al gene CCND1, che copre una regione tra i
marcatori D11S2663 e D11S4095e una seconda sonda (162kb) che coprel'estremità telomerica
del gene CCND1 e la regione fino al gene FGF3.
13q14.3 Deletion
Regione 13q14.2-q14.3, rosso (6.3-10.6ng/test)
Regione 13qter, 13q34 verde (24.7-36.9ng/test)
La sonda 13q14.3, marcata in rosso, copre i marcatori D13S319 e D13S25. La sonda
subtelomerica specifica per 13qter (clone 163C9), marcata in verde, consente l'identificazione
del cromosoma 13 e agisce come sonda di controllo.
Materiali forniti
Ogni kit contiene i seguenti reagenti, sufficienti per 2 (PMP 018), 5 (PMP 017) o 10 (PMP 016)
campioni del paziente:
1. 2, 5 o 10 dispositivi Chromoprobe Multiprobe®-CLL rivestito con sonde marcate
direttamente.
2. 4, 7 o 12 vetrini con impresso uno stampo specifico
3. 500µl di soluzione di ibridizzazione (formamide, destrano solfato, SSC)
4. 1 termometro di superficie per vetrini Cytocell
5. 1 camera di ibridizzazione Chromoprobe Multiprobe®Cytocell
Il kit per 20 test (PMP 020) è fornito come dispositivo Chromoprobe Multiprobe®4x5.
Avvertenze e misure precauzionali
1. Per uso diagnostico in vitro. Solo per uso professionale.
2. Indossare i guanti durante la manipolazione della soluzione di ibridazione, delle sonde di
DNA e della colorazione di contrasto DAPI.
3. La soluzione di ibridazione contiene formammide, che è un teratogeno; non inalare i fumi
ed evitare il contatto con la pelle. Indossare guanti, camice da laboratorio e manipolare in
una cappa aspirante. Per lo smaltimento, sciacquare abbondantemente con acqua.
4. DAPI è un potenziale cancerogeno. Maneggiare con cura; indossare guanti e camice da
laboratorio. Per lo smaltimento, sciacquare abbondantemente con acqua.
5. Lo smaltimento dei materiali pericolosi deve avvenire nel rispetto delle normative interne
relative allo smaltimento dei rifiuti pericolosi.
6. Gli operatori devono essere in grado di distinguere visivamente tra rosso, blu e verde.
7. Il mancato rispetto del protocollo può influire sulle prestazioni e generare risultati
falsi positivi o falsi negativi.
Conservazione e utilizzo
Il kit Chromoprobe Multiprobe®deve essere conservato a 2-8ºC fino alla data di scadenza
indicata sull’etichetta. Non congelare.
Attrezzature e materiali richiesti ma non in dotazione
1. 500µl di controcolorante (antifade DAPI (ES: 0,125µg/ml di DAPI (4,6-diamidino-2-
fenilindolo))).
2. Piastra riscaldante (con - un controllo accurato della temperatura fino ad 80ºC).
3. Micropipette a volume variabile compreso tra 1µl - 200µl.
4. Bagno termostatato con controllo accurato della temperatura a 72ºC.
5. Provette da microcentrifuga (0,5 ml).
6. Microscopio a fluorescenza (riferirsi alla sezione Configurazione ottimale del microscopio
e dei filtri).
7. Contenitori di Coplin in plastica o vetro.
8. Pinzette.
9. Olio per lenti ad immersione del microscopio a fluorescenza.
10. Centrifuga da banco.
11. Vetrini coprioggetto per microscopio a fluorescenza (24 x 50mm).
12. Timer.
13. Bagnomaria a 37ºC senza agitatore.
Configurazione ottimale del microscopio e dei filtri
Per una visualizzazione ottimale, utilizzare una lampada al mercurio da 100-W e obiettivi
apocromatici piani di tipo x63 o x100. Per una visualizzazione ottimale e simultanea dei fluorofori
rossi e verdi e DAPI, utilizzare un filtro passa banda triplo DAPI/FITC/Texas Red.
Prima dell’uso, verificare il corretto funzionamento del microscopio a fluorescenza. Utilizzare olio
da immersione adatto per microscopia a fluorescenza e formulato per bassa auto-fluorescenza.
Evitare di mischiare la soluzione DAPI Antifade con l’olio da immersione per microscopio poiché
questo oscura i segnali. Seguire le raccomandazioni dei produttori relative alla durata della
lampada e all’età dei filtri.
Preparazione del campione
La Chromoprobe Multiprobe®è concepita per essere utilizzata su cellule del sangue periferico
coltivate o su cellule del midollo spinale coltivate fissate nel fissativo di Carnoy che deve essere
preparato secondo le linee guida del laboratorio o dell'istituzione.
Preparare i campioni asciugati all’aria sui vetrini a stampo del Chromoprobe Multiprobe®Cytocell
in conformità con il protocollo Cytocell riportato qui sotto. Si sconsiglia di essiccare a caldo o
processare altrimenti i vetrini in quanto si potrebbe ridurre la fluorescenza del segnale.
Protocollo Chromoprobe Multiprobe®
N.B.: le sonde utilizzate con il dispositivo Chromoprobe Multiprobe®sono marcate direttamente
con fluorofori, che sono sensibili alla luce. Verificare che in ogni fase venga limitata l’esposizione
delle sonde alle luci del laboratorio (non è necessario lavorare al buio).
1. Preparazione del vetrino
a) Pulire un vetrino a stampo. Immergerlo per 2 minuti in metanolo al 100% e asciugarlo
minuziosamente con un panno morbido pulito.
b) Determinare l’indice mitotico corretto. È importante che i campioni designati abbiano un
indice mitotico sufficientemente alto da consentire la rilevazione della anomalie
cromosomiche. Per verificare la densità del campione, pipettare con una micropipetta (es.
una Gilson P10 o P20) 4µl della sospensione cellulare su una delle aree del vetrino a
stampo vuoto e lasciare asciugare all'aria. Vista l’entità minima del volume utilizzato, di
solito per trasferire la sospensione sarà necessario toccare delicatamente il vetrino con la
punta della pipetta. Esaminare al microscopio a contrasto di fase. Se la densità cellulare è
troppo elevata, diluire la sospensione con dell’altro fissativo. Se l’indice mitotico è troppo
basso, centrifugare a bassa velocità la sospensione cellulare fissata a 160 g per 10 minuti.